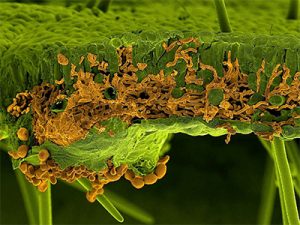
 Detalhe microscópico de uma folha de soja infectada pelo fungo Phakopsora pachyrhzi
Detalhe microscópico de uma folha de soja infectada pelo fungo Phakopsora pachyrhzi
O genoma altamente complexo do fungo P. pachyrhizi, causador da ferrugem asiática, a mais devastadora doença da soja, foi decifrado por um consórcio único de empresas públicas e privadas, do qual a UFV faz parte. A doença é o principal desafio para os produtores de soja no Brasil que, somente com custos de manejo, gastam mais de dois bilhões de dólares por safra. Se não for controlada, a ferrugem pode levar a perdas de até 90% de produtividade.
O anúncio da conclusão do sequenciamento do genoma do fungo foi feito dia 30 de setembro em escala mundial. O consórcio internacional é formado por representantes de 12 instituições públicas e privadas. Além da UFV, ele inclui a Fundação 2Blades, a Bayer, a Embrapa, as universidades alemãs de Hohenheim e de RWTH Aachen, o Instituto Nacional da Pesquisa Agronômica (INRA-França) e a Universidade de Lorraine (França). Também integra o consórcio o Joint Genome Institute (JGI, EUA), da KeyGene, do Laboratório Sainsbury (Reino Unido), da Syngenta.
Na UFV, o trabalho foi coordenado pelo professor Sérgio Brommonschenkel, do Laboratório de Genética e Genômica de Interações Planta/Patógeno. Os pesquisadores envolvidos concluíram o sequenciamento e a montagem não apenas do genoma de um, mas de três isolados de P. pachyrhizi.
Segundo o professor Brommonschenkel, o patógeno é difícil de ser combatido porque é capaz de se adaptar às inúmeras estratégias de controle já conhecidas. Para acelerar a inovação, desenvolver novas características e descobrir novos modos de ação contra o fungo era essencial decifrar o genoma considerado de alta complexidade, o que dificultava a sua caracterização pelas técnicas tradicionais de sequenciamento.
Para se ter uma ideia da complexidade, a montagem do genoma de referência abrange 1,057 Gb, por tratar-se de um dos maiores genomas entre fungos e patógenos vegetais em geral. Apesar de seu tamanho, o número de genes preditos em seu genoma é de cerca de 20.700, o que é semelhante ao número encontrado em outros fungos da ferrugem. O consórcio também gerou um atlas de expressão (transcriptoma) de todas as estruturas fúngicas em diferentes estágios de infecção do patógeno. Esse conhecimento, segundo o professor Brommonschenkel, contribuirá para melhorar a anotação e ampliar o conhecimento dos mecanismos moleculares explorados por P. pachyrhizi durante o processo de patogênese.
O genoma de outros dois isolados do fungo foram sequenciados e montados na UFV, pelo grupo do Laboratório de Genética e Genômica de Interações Planta/Patógeno. “A disponibilidade de um genoma de referência oferece uma oportunidade única para entender os mecanismos genéticos empregados pelo fungo para quebrar a resistência de variedades comerciais e também desenvolver resistência aos fungicidas”, explica o professor. Para ele, o conhecimento detalhado do arsenal genético empregado pelo patógeno em sua interação com a soja e outros hospedeiros pode contribuir para o desenvolvimento de estratégias inovadoras e disruptivas para o controle desse importante patógeno.
Todos os dados do sequenciamento genético estão disponíveis para a comunidade científica no link https://mycocosm.jgi.doe.gov/Phapa1